Questa famiglia di virus ha assunto protagonismo nel corso della pandemia della Covid-19 e precedentemente nelle epidemie di SARS e MERS.
I Coronavirus rappresentano la seconda causa del raffreddore comune negli umani dopo i Rhinovirus. Questo però non significa che la patogenicità di tutti i virioni di questo gruppo sia clinicamente così poco rilevante. Alcuni virus della famiglia dei Coronavirus (CoV) che infettano altre specie animali, presentano infatti diversi gradi di neurovirulenza. Nel maiale ad esempio, una grave malattia cerebrale, l’encefalomielite emaglutinante, ha come agente eziologico un virus che presenta il 91% di omologia (ovvero somiglianza genetica) con un coronavirus umano l’OC43.[1]
Anche gatti e topi possono essere infettati da Coronavirus neuroinvasivi e sebbene non siano delle zoonosi e pertanto non costituiscano -ad oggi- alcun pericolo per l’uomo, il fatto che alcuni CoV possano mostrare una certa neurovirulenza, ha fatto scattare l’attenzione verso il Sars-CoV-2 umano. De facto, vi furono episodi di encefalomielite da MERS [2] ma le infezioni primarie del SNC da Coronavirus sembrano essere infrequenti.
Il Sars-CoV-2 è un virus a RNA appartenente, come sappiamo, ai Coronaviridae (CoV). Di questa famiglia di virioni, caratterizzati da una spiccata capacità ricombinate, il Sars-CoV-2 si aggiunge ad altri sei in grado di infettare l’organismo umano:
- Alfacoronavirus 229E (HCov-229E)
- Alfacoronavirus NL63 (HCoVNL63)
- Betacoronavirus HKU1 (HCoV-HKU1)
- Betacoronavirus OC43 (HCoV-OC43)
- MERS-Cov6
- Sars-Cov
I primi quattro sono endemici a livello mondiale e costituiscono appunto l’agente eziologico nel 10% dei casi di raffreddore comune, ma i CoV umani sono entrati con prepotenza sulla scena mondiale agli inizi del XXI secolo.
la SARS
Il 15 marzo del 2003 l’OMS allertava per la presenza di focolai infettivi di una polmonite atipica nella provincia cinese di Guangdong, il cui patogeno fu poi identificato come un CoV. La malattia presentava una patogenesi prevalentemente a carico delle vie respiratorie, da cui il nome SARS (Severe Acute Respiratory Syndrome). Il virus si diffuse in 32 Stati ma il focolaio infettivo più importante al di fuori dei confini cinesi, si ebbe in Canada nella città di Toronto. Non fu chiaro sin da subito quale potesse essere il serbatoio dell’infezione. Inizialmente si pensò allo zibetto quale reservoir naturale, ma ben presto si capì che questo piccolo animale sviluppando lui stesso la malattia, ne costituiva un semplice ospite intermedio. Posteriormente si identificò in una specie di pipistrello (il Rhinolophus) l’ospite naturale del patogeno. Il tasso di letalità fu del 10,9% [3].
l genoma virale codifica quattro proteine strutturali:
- la proteina E dell’envelope,
- la M della membrana,
- la N del nucleocapside
- e le assai note già citate S (spikes) che si trovano sulla superficie virale e che conferiscono il caratteristico aspetto a corona del germe osservato al microscopio elettronico.
La Spike-protein ha due sub-unità, la sub-unità S1 è responsabile del tropismo virale, mentre la sub-unità S2 è coinvolta nella fusione con le cellule dell’ospite ed è in grado di legarsi al recettore angiotensin-converting enzime 2, conosciuto come ACE2, che si trova negli pneumociti, nelle cellule dell’endotelio vascolare, del muscolo liscio e nei reni. Si deduce, quindi, che il quadro clinico interessa prevalentemente questi distretti in special modo le vie respiratorie con un effetto citopatico sulle cellule polmonari e manifestazioni respiratorie anche gravi.
la MERS
Dieci anni dopo si presentava una nuova epidemia, questa volta con un tasso di letalità ben più alto della precedente, del 34,4% [4]. La malattia si estese nel 2012 ed ebbe come principale area la Giordania e l’Arabia Saudita, ovvero il Medioriente, da cui il nome MERS (Middle East Respiratory Syndrome) ma nel 2015 si verificò un importante focolaio in Corea del Sud. Nell’ecologia del virus anche in questo caso si identificarono i pipistrelli (Pipistrellus e Perimyotis) come reservoir naturali ed i cammelli e dromedari come ospiti intermedi.
Ricombinazione virale
Le alte capacità ricombinanti di questi virus e gli studi filogenetici avvallano l’ipotesi che il Sars-CoV-2 abbia fatto anch’esso un salto di specie. La sequenza genomica del Sars-CoV-2 fu resa pubblica dalle autorità cinesi nel database GenBank (MN908947) e presenta una omologia del 96,2% con il Coronavirus BatCoV RaTG13 (vedi immagine sotto) isolato in un pipistrello nella provincia dello Yunnan e dell’82% col SARS-CoV. È ormai parere condiviso nella comunità scientifica internazionale, identificare negli allevamenti misti suini e avicoli, e nei più conosciuti “wet markets” i luoghi privilegiati dove ceppi virali diversi di specie animali diverse possono ricombinarsi geneticamente e favorire gli infausti spillover.
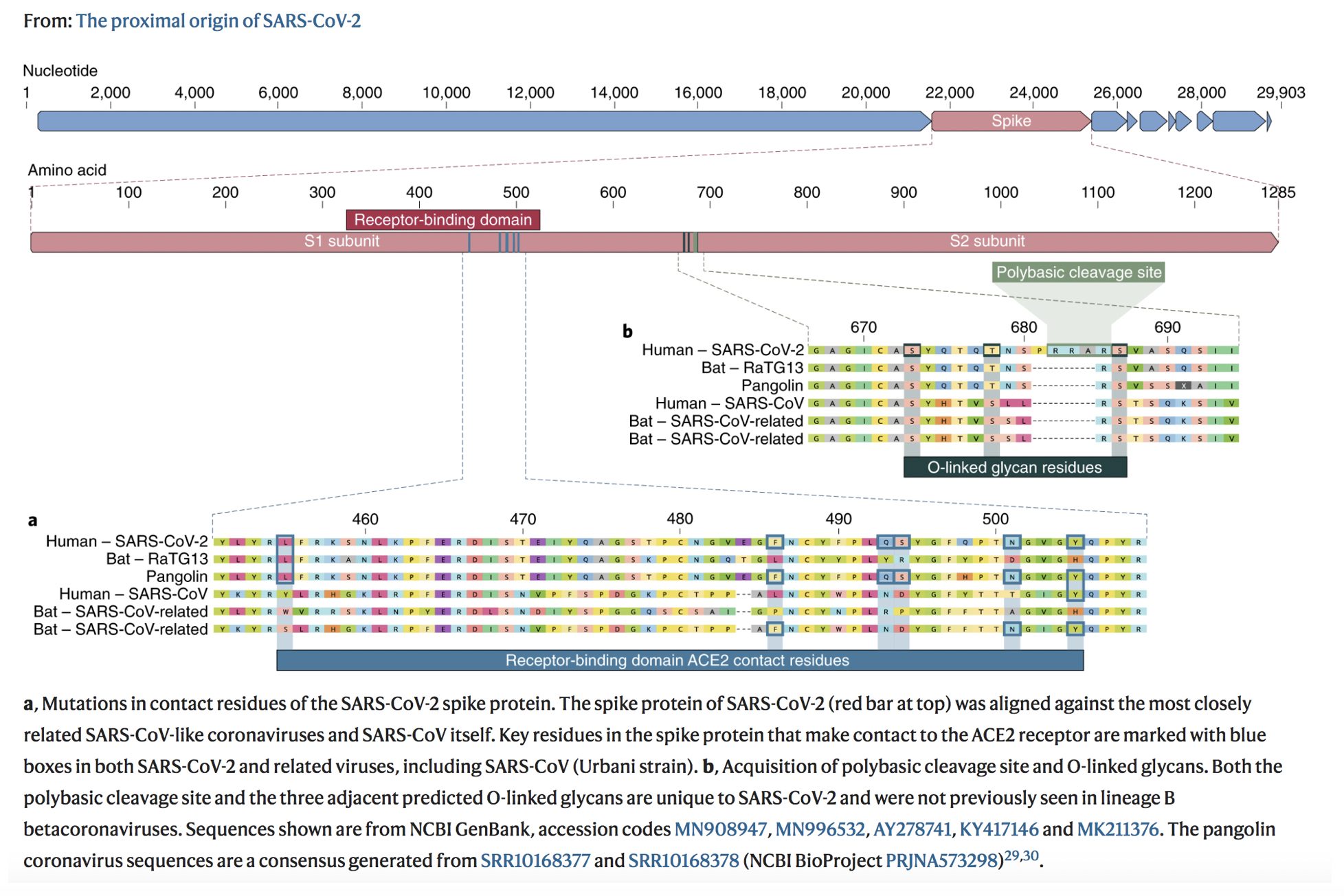
Se si osservano le prime due linee accanto alla lettera “a” (quella in alto del Sars-CoV-2 umano e quella poco più in basso del Coronavirus del pipistrello, il Bat-RaTG13), si evince con chiarezza la somiglianza nell’intera sequenza genetica della proteina Spike tra i due virus.
[1] Francisco J. Carod-Artral, Complicaciones neurológicas por coronavirus y Covid-19. Revisión. Rev Neurol 2020, 70 (9): 311-322.
[2] Arabi YM, Harthi A, Hussein J, Bouchama A, Johani S, Hajeer AH, et. Al. Severe neurologic síndrome associated with Middle East respiratory síndrome corona virus (MERS-CoV). Infection 2015; 43:495-501.
[3] Mauro Moroni, Spinello Antinori, Claudio Maria Mastroianni, Vincenzo Vullo. Manuale di malattie infettive. 3ª edizione, 2020 Edra.
[4] Ibidem.
[5] Han H, Xu Z, Cheng X, et al. Descriptive, retrospective study of the clinical characteristics of asymptomatic COVID-19 patients. mSphera, 2020 oct 7;5(5).
[6] Arcanjo, A., Logullo, J., Menezes, C.C.B. et al. The emerging role of neutrophil extracellular traps in severe acute respiratory syndrome coronavirus 2 (COVID-19). Sci Rep 10, 19630 (2020). https://doi.org/10.1038/s41598-020-76781-0.
[7] https://www.semg.es/index.php/noticias/item/613-noticia-20201111.
[8] Venturelli, S., Benatti, S., Casati, M., Binda, F., Zuglian, G., Imeri, G., . . . Rizzi, M. (2021). Surviving COVID-19 in Bergamo Province: A post-acute outpatient re-evaluation. Epidemiology and Infection,1-25. doi:10.1017/S0950268821000145.
[9] IES-R (Impact of Events Scale – Revised): la Scala di impatto dell’evento è uno strumento classico nella valutazione dei disturbi post-traumatici; HADS (Hospital Anxiety and Depression Scale): è un questionario di facile somministrazione che valuta lo stato di sofferenza psichica in pazienti ricoverati in reparti ospedalieri ed è costituito da 14 item; RSA (Resilience Scale for Adults): è un questionario di 33 item con buone proprietà psicometriche. Indaga sei dimensioni psichiche: Percezione di sé; Fiducia nella possibilità di pianificare il futuro; Competenze sociali; Orientamento agli obiettivi e all’organizzazione della vita quotidiana; Coesione familiare; Risorse sociali; MoCA (Montreal Cognitive Assessment): è uno degli strumenti più utilizzati per la valutazione neuropsicologica: Indaga le seguenti aree funzionali: attenzione, concentrazione, funzioni esecutive, memoria, linguaggio, abilità visuo-costruttive, astrazione, calcolo, orientamento. Relativamente ai disturbi cognitivi il Montreal Cognitive Assessment è risultato positivo solo in due pazienti, sebbene 69 riferissero di presentare disturbi della sfera cognitiva.
[10] Elizabeth M. Rhea, Aric F. Logsdon, Kim M. Hansen, Lindsey M. Williams, May J. Reed, Kristen K. Baumann, Sarah J. Holden, Jacob Raber, William A. Banks, Michelle A. Erickson. The S1 protein of SARS-CoV-2 crosses the blood–brain barrier in mice. Nature Neuroscience, 2020; DOI: 10.1038/s41593-020-00771-8.
[11]Eugenia Martínez Hernandéz, Roser Velasco Fargas. Mecanismos fisiopatogéneticos de afectación neurológica. In: David Ezpeleta, David Garcia Azorín, Manual Covid-19 para el neurólogo general. Ediciones SEN, 2020.